Introduction aux serveurs d'exploration sous Unix : Différence entre versions
imported>Jacques Ducloy (→HfdCat) |
imported>Jacques Ducloy (→HfdCat) |
||
| Ligne 168 : | Ligne 168 : | ||
HfdCat Data/Main/Exploration/AffPays.i.hfd | SxmlIndent | more | HfdCat Data/Main/Exploration/AffPays.i.hfd | SxmlIndent | more | ||
</source> | </source> | ||
| + | ===SxmlSelect=== | ||
| + | La commande [[SxmlSelect (Dilib)|SxmlSelect]] vue précédemment va naturellement s'appliquer sur les ensembles Hfd. | ||
Version du 18 juin 2020 à 09:53
|
Introduction aux serveurs d'exploration sous Unix < = Introduction à la programmation en C des arbres Xml |
Ce module donne des premiers éléments pour l'exploration de serveurs d'exploration sous Unix.
Les exemples seront donnés sur le Serveur d'exploration sur la grippe en Belgique.
Sommaire
Organisation HFD
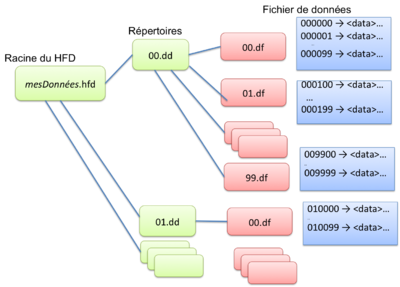
Les serveurs d'exploration utilisent une organisation de fichiers spécifique qui a pour nom : HFD ( Hierarchical File organisation for Documentation ).
Principe
Ils gèrent des collections de documents XML et plus précisément sur une ligne physique (Stream XML).
Chaque document est identifié par une clé séquentielle de longueur fixe (6 caractères). En notation décimale il est possible de ranger 1 000 000 objets.
Dans ce cas, à chaque document Sxml est associé une clé à 6 chiffres (de 000000 à 999999).
- les 2 premiers chiffres repèrent un répertoire (suffixé par dd),
- les 2 chiffres du milieu repèrent un fichier (suffixé par df),
- les 3 derniers repèrent le document dans le fichier (en fait chaque document Sxml est précédé par la clé complète).
Par exemple, la document 123456 sera :
- le 57ème document Sxml
- d'un fichier nommé 34.df
- rangé dans le répertoire 12.dd
Son adresse dans le système de fichier Unix sera alors :
- maBiblio.hfd/12.dd/34.df
En fait de façon implicite, l'organisation repose sur des clés hexadécimales.
Première exploration
Pour le serveur GrippeBelgiqueV2, la plateforme Unix d'exploration est située ici :
$WICRI_ROOT/Sante/explor/GrippeBelgique.storage/GrippeBelgiqueV2
Observer le résultat des commandes suivantes :
cd $WICRI_ROOT/Sante/explor/GrippeBelgique.storage/GrippeBelgiqueV2
ls
ls Data
ls Data/Main
ls Data/Main/Exploration
ls Data/Main/Exploration/biblio.hfd
ls Data/Main/Exploration/biblio.hfd/00.dd
ls Data/Main/Exploration/biblio.hfd/00.dd/00.df
more Data/Main/Exploration/biblio.hfd/00.dd/00.df
cat Data/Main/Exploration/biblio.hfd/00.dd/00.df | SxmlIndent | more
ls Data/Main/Exploration/Author.i.hfd/00.dd/
head -5 Data/Main/Exploration/Author.i.hfd/00.dd/00.df
ls Data/Main/Exploration/AbsEn.i.hfd/00.dd/
head -5 Data/Main/Exploration/AbsEn.i.hfd/00.dd/00.df
Organisation d'une plateforme d'exploration et de curation
L'expression serveur d'exploration désigne un ensemble de procédures qui permettent d'explorer un corpus, généralement composé de textes scientifiques avec des métadonnées structurées.
En réalité l'approche ScienceExplor propose un ensemble de serveurs d'exploration pour travailler sur un ensemble de corpus à différents stages de transformation.
Organisation physique
A la racine d'un serveur, la commande précédente ls montre un ensemble de répertoires :
- GrippeBelgiqueV2 -+ $EXPLOR_AREA
+- Data
+- Import
+- Input
+- Make
+- Site
+- bin
Le répertoire Data
Le répertoire Data, dans les cas simples, (hors ISTEX), contient l'ensemble des données dans leur forme exploratoire (en XML, autour de la DTD TEI, avec des homogénéisations de vocabulaires).
Il est organisé en flux (stream) et étapes (step).
Dans le cas d'un serveur monosource (ici PubMed) il y a un seul flux nommé Main.
Ce flux est organisé en 3 étapes : Corpus, Curation, Exploration.
Pour chaque étape, on trouve un ensemble de « fichiers HFD » :
- un fichier
biblio.hfdqui contient les documents dans leurs différents stades d'élaboration. - des fichiers d'index (fichiers inverses), exemple
AffPays.i.hfd.
Le répertoire Site
Ce répertoire Data peut être exploré sous Unix, ou par les fonction php qui sont regroupées dans le répertoire Site.
Le répertoire Site est organisé dans la perspective d'une version multilingue.
En faisant « ls Site/fr/Main » vous retrouvez (entre autres choses) des répertoires liés aux étapes.
Vous pouvez comparer :
- La page d'accueil sur le site lorexplor
- le fichier Site/fr/index.html
Ou encore (observer également l'url) :
- La page d'accueil de l'étape Exploration
- le fichier Site/Main/Exploration/fr/index.html
Le répertoire Import
Il contient les données brutes :
- les téléchargements de corpus (ici pubmed_result.xml),
- des copies de pages de paramètres, comparez :
- wicri-outils.fr:Wicri:ExplorPubmed/Parameter data
- le fichier :
WicriAreaParam.data.wiki
Les répertoires bin, Make et Input
Ces répertoires ont une fonction très techniques. Ils sont créés par le programme de génération.
Le répertoire bin contient un ensemble de commandes shell. Voir :
ls bin cat bin/MainCreateCorpus.sh cat bin/MainCorpusBiblio.sh
La génération d'une plateforme est pilotée par un « makefile » qui définit les relations de dépendance entre les actions. Il sera utilisé par une commande unix nommée make qui lance les actions en fonction de ces dépendances.
Si bous visualisez ce fichier (cat bin/area.mk), vous verrez par exemple :
$(EXPLOR_AREA)/Make/Main.stream: \
$(EXPLOR_AREA)/Make/Main/Corpus.step \
$(EXPLOR_AREA)/Make/Main/Curation.step \
$(EXPLOR_AREA)/Make/Main/Exploration.step
touch $(EXPLOR_AREA)/Make/Main.streamCela signifie : pour fabriquer les données du flux Main, il faut fabriquer les 3 étapes Corpus, Curation et Exploration. Puis signaler la fin de ce travail avec une commande touch.
Plus loin, on trouve :
$(EXPLOR_AREA)/Make/Main/Corpus.step: \
$(EXPLOR_AREA)/Make/Main/Corpus/biblio \
...
$(EXPLOR_AREA)/Make/Main/Corpus/AutAff.i.index
touch $(EXPLOR_AREA)/Make/Main/Corpus.stepCela signifie pour faire l'étape Corpus, il faut fabriquer la bibliographie et des fichiers inverses.
Puis :
GrippeBelgiqueV2/Make/Main/Corpus/biblio: \
GrippeBelgiqueV2/Import/pubmed_result.xml
sh GrippeBelgiqueV2/bin/MainCorpusBiblio.sh
touch GrippeBelgiqueV2/Make/Main/Corpus/biblioLa biblographie dépend du fichier pubmed_result.xml (plus précisément s'il est modifié, il faut re-générer le serveur). Pour cela, exécuter MainCorpusBiblio.sh </sh>. Puis signaler que cela s'est bien passé avec un touch.
Le répertoire Make contient les fichiers de synchronisation créés par les commandes touch.
La répertoire Input contient des tables de paramètres, codées en xml.
Quelques commandes sur les organisations HFD
La bibliothèque Dilib contient un ensemble de commandes pour manipuler les organisations Hfd.
HfdCat
HfdCat est le nom d'une commande qui permet de d'accéder séquentiellement (comme un cat) à l'ensemble des documents d'un fichier HFD.
On peut ainsi atteindre tous les documents d'un corpus et leur appliquer des commandes.
Par exemple, pour voir l'effet de la curation, si l'on veut savoir combien de documents contiennent les mots Brussels ou Bruxelles, il faut faire :
HfdCat Data/Main/Exploration/biblio.hfd | grep Brussels | wc
HfdCat Data/Main/Corpus/biblio.hfd | grep Brussels | wc
HfdCat Data/Main/Corpus/biblio.hfd | grep Bruxelles | wc
HfdCat Data/Main/Exploration/biblio.hfd | grep Bruxelles | wc
Pour explorer un fichier inverse, on peut faire :
HfdCat Data/Main/Exploration/AffPays.i.hfd | SxmlIndent | more
SxmlSelect
La commande SxmlSelect vue précédemment va naturellement s'appliquer sur les ensembles Hfd.